HiBiT Technology
HiBiT Technology
テクノロジー概要
- HiBiTシステムとは
-
HiBiT system は11アミノ酸のペプチドタグ(HiBiT)と、それに結合する約18kDa のNanoLuc® ルシフェラーゼ断片(LgBiT)と基質を用いた、発光法によって目的タンパク質を検出する技術です。
すでに実用化されているタンパク質検出タグとして、アフィニティータグ(His、c-Myc、FLAG 等)やGFP 等の蛍光タンパク質が知られていますが、簡便性、親和性、アプリケーション、ランニングコストの面で長所と短所があるため、実験により使い分ける必要がありました。HiBiT system はタンパク質定量に関して、これらいずれの点においても高い性能を持ち、これまで難しいとされた様々な実験系を実現できる世界初の新規発光タグシステムです。
- 生細胞で “生きたまま” タンパク質定量(生細胞アッセイ)、溶液中やブロッティングメンブレン上のタンパク質定量(細胞溶解アッセイ)が可能
- 抗体不要、試薬を加えるだけの簡単、迅速アッセイ
- わずか11アミノ酸のタグは、ウイルスゲノム挿入やゲノム編集に最適
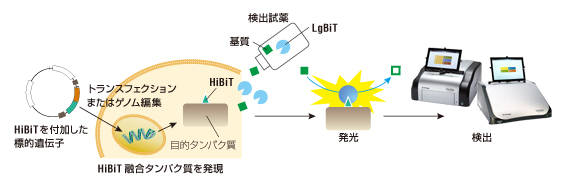
HiBiTシステムの基本的フロー
*LgBiTは細胞膜を透過しないため、細胞内部のHiBiTを検出する際には細胞溶解が必要となります。
- HiBiT開発までの道のり
-
2012年、プロメガはより明るく小さい新規のルシフェラーゼとしてNanoLuc® ルシフェラーゼを開発しました。これにより従来のレポーター遺伝子アッセイの高感度化だけでなく、タンパク質レポーターとして新たなアプリケーションにも展開できるようになりました。
2015年、大小2つのNanoLuc® 断片の相補性を利用したNanoBiT® System をタンパク質間相互作用の解析ツールとして開発しました。この技術により、これまで難しかった生細胞でのタンパク質間相互作用を高感度に定量できるようになりました。開発の過程で大きな断片であるLarge BiT(LgBiT)[ 約18kDa] と相補性を持つ小さなペプチド断片(11アミノ酸)をスクリーニングし、最も親和性の低い断片(Kd=190 μM)をSmall BiT(SmBiT)としてNanoBiT® に採用しました。さらに、最も高い親和性を有するペプチド(Kd=700 pM)をHiBiT と名づけ、発現タンパク質を高感度に検出するための発光タグとして様々なアプリケーションについて検討を行いました。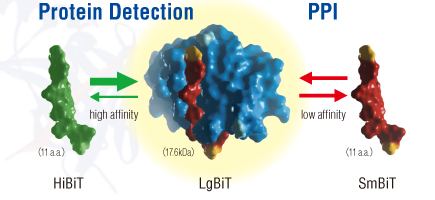
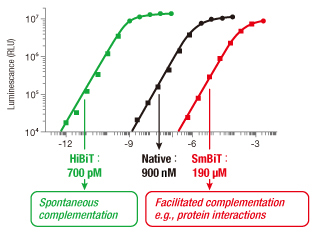
スクリーニングより得られたペプチドとLbBiTとの親和性
- アルファテスターのご協力
-
HiBiT は不可能を可能にする新しいタンパク質検出方法として広範な用途への利用が考えられ、多く の方にテスターとしてご意見を賜りました。日本国内でも30名以上のテスターの方に種々のテスト実験をして頂き、頂いたフィードバックをもとに製品開発の参考にさせて頂きました。そのうちの3名のテスター参加者への導入エピソードをご紹介致します。
ハイスループットかつ”生きたまま”の分泌タンパク質定量(HiBiT Extracellular Detection)東京医科歯科大学大学院医歯学総合研究科 システム発生・再生医学研究分野 松島隆英 先生
松島先生はこれまで分泌タンパク質の細胞外での定量に様々な手法を検討されていました。ELISA assay や質量分析の手法では相性のよい抗体が必要、また HTSに向かないなどの問題がありました。そこで、NanoLuc® 融合タンパク質を用いた実験系をご紹介し、特に HTSでの簡便な実験系ができるようになりました。さらに今回 HiBiT をご紹介し、より内在的な解析のための CRISPR/Cas9 system によるゲノム編集が 可能になり、培養細胞だけでなく最終的には究極的な”生きたまま”の実験系であるマウス in vivo解析へと展開して頂いています。
レポーターのウイルスゲノムへの導入(HiBiT Intracellular Detection)大阪大学微生物病研究所 分子ウイルス分野 福原崇介 先生
福原先生はハイスループットなスクリーニング系構築のための、様々なウイルスを利用した変異ウイルスレ ポーター作製を研究しておられました。問題点として、小型ウイルスでは Firefly などの大きなルシフェラーゼ の挿入は困難であるという点がありました。そんな中、小型のレポーターとして NanoLuc® をご使用頂き、C 型肝炎ウイルスなどの小型ウイルスへの導入が可能であることを検証頂いておりました。さらに今回、ペプチ ドレベルまで最小化したHiBiTをご紹介し、これまでレポーターの挿入が不可能であった多くのウイルスにレ ポーターの導入が可能になるとの声を頂き、検証して頂いております。
筋芽細胞の細胞融合の定量化(HiBiT Intracellular Detection)名古屋大学 医学部保健学科 理学療法学専攻 亀高 諭 先生
亀高先生は骨格筋が再生する際に見られる筋芽細胞の融合現象の定量化の手法を探しておられました。細胞融合のモニタリングにはこれまでGFPやRFPなどの蛍光タンパク質が一般的でした。2つの細胞にそれぞれ の蛍光タンパク質を発現させ、細胞融合によりそれらがmergeし、ダブルポジティブな細胞をイメージング により細胞融合を評価する系です。ただしこの手法は基本的にはイメージングでの定量であるため、定量性 やスループットに限界がありました。今回 HiBiT をご紹介したところ、細胞融合により発光定量ができる点に 注目頂き、これまで実現できなかった細胞融合の定量化手法としてご検討頂くことになりました。
HiBiT 配列の導入
- 標的遺伝子にHiBiT配列を付加する
-
HiBiT 配列は33塩基にコードされる極小のペプチドタグ(11 アミノ酸)なので、従来法のようにHiBiT ベクターへ目的遺伝子をクローニングするだけでなく、短いエピトープタグなどと同様にお手元の標的遺伝子発現クローンへPCR 法や遺伝子合成により簡便に付加することもできます。また、CRISPR/Cas9 などのゲノム編集技術により内在遺伝子に融合させることも可能です。
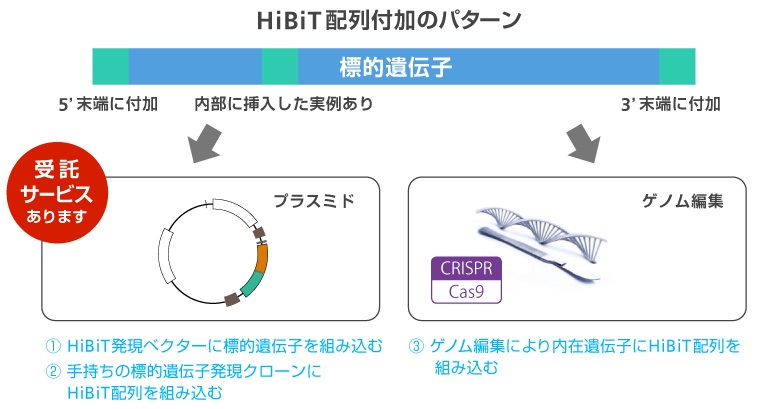
- ベクターの種類
-
HiBiT配列を含むベクターは、標的タンパク質のN末端に付加するタイプ、C末端に付加するタイプ、分泌シグナルつきタイプから選べます。さらにそれぞれマルチクローニングサイトを含むもの、Flexi® クローニングが可能なものがあります。
これらベクターに、標的遺伝子のDNA断片を組み込むいくつかの方法を以下にご紹介しますが、標的遺伝子のDNA断片をフレームを合わせてベクターに挿入できればどのような方法でも良く、適当な制限酵素サイトの有無や慣れ、好みなどにより実施しやすい方法で行っていただけます。
HiBiT ベクター(MCS クローニング)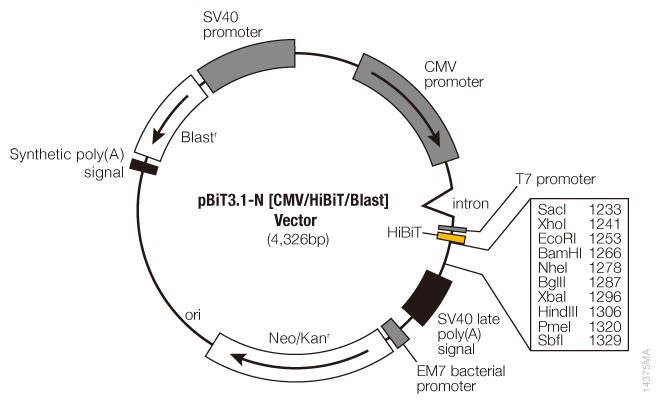
横スクロールできます→
製品名 カタログ番号 HiBiTが付加される部位 HiBiT ベクター(MCS クローニング) pBiT3.1-N[CMV/HiBiT/Blast ]Vector N2361 N末端 pBiT3.1-C[CMV/HiBiT/Blast ]Vector N2371 C末端 pBiT3.1-secN[CMV/HiBiT/Blast ]Vector N2381 N末端、IL-6分泌シグナルつき HiBiT ベクター(Flexi® クローニング) pFN38K HiBiT CMV-neo Flexi® Vector N2401 N末端 pFC37K HiBiT CMV-neo Flexi® Vector N2391 C末端 pFN39K secHiBiT CMV-neo Flexi® Vector N2411 N末端、IL-6分泌シグナルつき 横スクロールできます→
-
HiBiTベクターをご使用の際には、営利企業、非営利団体を問わずラベルライセンスの内容を承認いただく必要があります。製品を開封するとラベルライセンスに同意したことになります(その他手続きは必要ありません)。
また、HiBiTの配列をPCRなどで合成する場合、ラベルライセンスの内容を承認後、ご登録が必要となります(1分で終了する配列利用に関する簡単な登録です)。
詳しくはこちら
- 1. HiBiT発現ベクターに標的遺伝子を組み込む
-
- 受託サービスあります
- クローニングビギナー向け
- 受託サービスをフル活用の最も簡単な方法
こんな方に:標的遺伝子(ヒト遺伝子)が手元になく、クローニング作業はすべて省略したい方
- 標的遺伝子をクローンコレクションから選択して購入
(Flexi® ORF Clone:かずさDNA研究所保有の約20,000のヒト遺伝子コレクション) - HiBiT Flexi® Vector(カタログ番号 N2391, N2401, N2411)を購入
- サブクローニング受託サービスに申込み
(ご購入いただいたクローンに含まれる標的遺伝子をHiBiT発現ベクターにサブクローニング)
- 標的遺伝子をクローンコレクションから選択して購入
- Flexi® クローニング(Flexi® OFRクローンを利用する場合)
こんな方に:標的遺伝子(ヒト遺伝子)は購入してクローニングの手間を省き、サブクローニングはコストセーブのため自分で行いたい方
- 標的遺伝子をクローンコレクションから選択して購入
(Flexi® ORF Clone:かずさDNA研究所保有の約20,000のヒト遺伝子コレクション) - HiBiT Flexi® Vector(カタログ番号 N2391, N2401, N2411)を購入
- Flexi® Cloning System(カタログ番号:C8640, C9320, C8820)でFlexi クローニング(N末端 and/or C 末端付加)
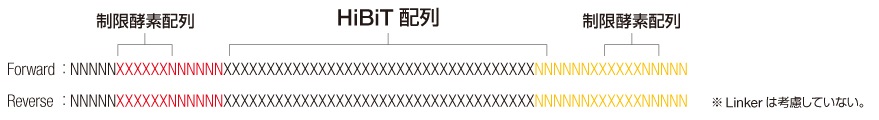
*リンカーは下図の配列が付加されます。
POI: protein of interest横スクロールできます→
ベクター名 融合タンパク質 pFC37K HiBiT CMV-neo Flexi® Vector POI-VSQGSSGGSSG-HiBiT pFN38K HiBiT CMV-neo Flexi® Vector HiBiT-GSSGGSSGAIA-POI pFN39K secHiBiT CMV-neo Flexi® Vector IL6-HiBiT-GSSGGSSGAIA-POI 横スクロールできます→
- 標的遺伝子をクローンコレクションから選択して購入
- MCS クローニング(ご自身で準備された遺伝子をマルチクローニングサイト[MCS]より 挿入される場合)
こんな方に:標的遺伝子ORFのDNA断片をお持ちの方、ヒト以外の遺伝子を使用したい方
- 標的遺伝子クローンやcDNA を準備
- PCR and/or 制限酵素処理により標的遺伝子ORF を単離
*下図の制限酵素サイト、リンカー配列を参照 - Wizard® SV Gel and PCR Clean-Up System(カタログ番号:A9281)で精製
- HiBiT MCS Vector(カタログ番号:N2361, N2371, N2381)とORF をLigaFast™ Rapid DNA Ligation System(カタログ番号:M8221, M8225)ライゲーション、セレクション
- HiBiT 付加遺伝子発現クローン完成
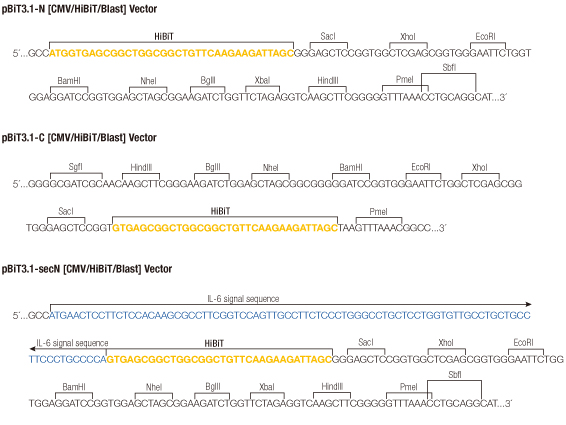
pBiT3.1 Vector のMCSで利用できる制限酵素サイト横スクロールできます→
ベクター名 融合タンパク質 MCS 制限酵素サイト pBiT3.1-N [CMV/HiBiT/Blast] Vector HiBiT-GSSG-POI SacI HiBiT-GSSGGSSG-POI XhoI HiBiT-GSSGGSSGGNS-POI EcoRI HiBiT-GSSGGSSGGNSGGGS-POI BamHI pBiT3.1-C [CMV/HiBiT/Blast] Vector POI-GSSG-HiBiT SacI POI-GSSGGSSG-HiBiT XhoI POI-GNSGSSGGSSG-HiBiT EcoRI POI-GSGGNSGSSGGSSG-HiBiT BamHI pBiT3.1-secN [CMV/HiBiT/Blast] Vector IL6-HiBiT-GSSG-POI SacI IL6-HiBiT-GSSGGSSG-POI XhoI IL6-HiBiT-GSSGGSSGGNS-POI EcoRI IL6-HiBiT-GSSGGSSGGNSGGGS-POI BamHI 横スクロールできます→
- 2. 手持ちの標的遺伝子発現クローンにHiBiT配列を組み込む
-
- クローニングに慣れている方向け
HiBiT配列を合成し、標的遺伝子発現ベクターに挿入する方法です。
- 受託サービスをフル活用の最も簡単な方法
こんな方に:標的遺伝子(ヒト遺伝子)が手元になく、クローニング作業はすべて省略したい方
- ライセンス承認登録
ご登録後、塩基配列ならびにアミノ酸配列を記したメールをお送りしております。 - 手持ちの目的遺伝子発現ベクターへのHiBiTの挿入位置を決定
- インサート作製用oligoのデザインおよび発注
(oligoのデザイン例)
- oligoをアニーリング、またはPCR増幅する
- ベクターおよびインサートを制限酵素処理し、Wizard® SV Gel and PCR Clean-Up System(カタログ番号:A9281)で精製
- LigaFast™ Rapid DNA Ligation System(カタログ番号:M8221, M8225)でベクターにHiBiTの配列を組み込む
- 組み替えたベクターを大腸菌にトランスフォーメーション
- HiBiT付加遺伝子発現クローン完成
- 3. ゲノム編集により内在遺伝子にHiBiT配列を組み込む
-
- 内在レベルの発現を希望する方向け
CRISPR/Cas9ゲノム編集によるHiBiTノックインの方法です。
- ライセンス承認登録
ご登録後、塩基配列ならびにアミノ酸配列を記したメールをお送りしております。 - ゲノム上のターゲット配列確認
- crRNA のデザインおよびガイドRNA(crRNA + tracrRNA)のデザインおよび発注
- HiBiT Donor DNA template のデザインおよび発注
アプリケーション
- HiBiTの特徴
-
HiBiTシステムのベースとなるHiBiTは大きさがわずか11アミノ酸であり、高い親和性(Kd = 約1 nM)でLgBiTと結合してNanoLuc® を再構成し、強い発光を生じます。この非常に明るい光により過剰発現させることなく内在レベルで発現する目的タンパク質の定量化が可能であり、CRISPR等のゲノム編集技術と組み合わせて内在ローカスにこのHiBiTタグを埋め込むことで実質的にあらゆるタンパク質の発現をモニタリングすることができます。また、SDS-PAGEで分離したHiBiTタグ付きタンパク質をブロッティングし、LgBiTを含む検出試薬を添加するだけでフェムトグラムレベルの感度でタンパク質を検出することがきます。さらに、細胞を溶解することなくHiBiTタグタンパク質の細胞表面発現、膜受容体の内在化、また分泌タンパク質を数分で測定することもできます。シンプルな1回試薬添加のアッセイプロトコールなのでハイスループットアプリケーションにも最適です。
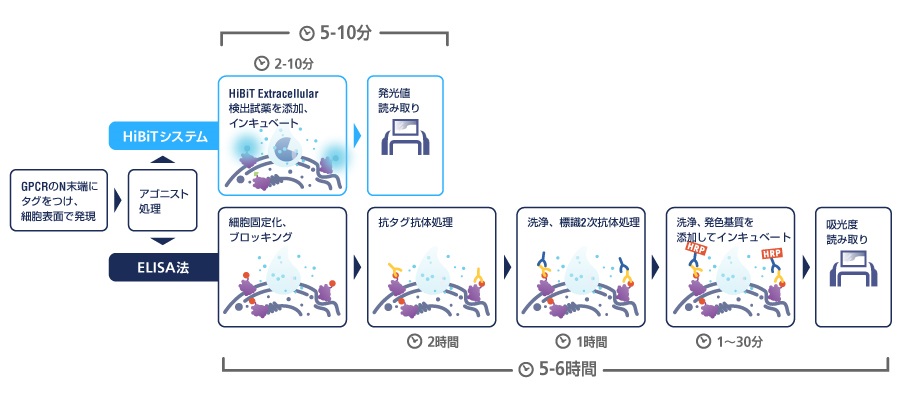
HiBiTシステムとELISA法の比較例
細胞表面に露出したGPCRを検出した事例より(詳細はこちら)
- アプリケーション
-
発現タンパク質の定量
細胞ライセートのタンパク質定量 <HiBiT Lytic Detection>

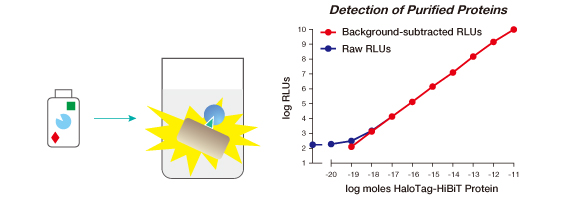
目的タンパク質にHiBiTを付加し、細胞内で発現させ、細胞溶解成分を含む試薬(LgBiT、NanoLuc® 基質を供給)を添加して発光測定する。
- 試薬を1回添加するだけのシンプルな手順
- 10分以内に実験完了
- 7桁以上の広いダイナミックレンジ(30kDaタンパク質で10fg-100ng)
-
ブロッティングメンブレン上でのタンパク質定量 <HiBiT Blotting>
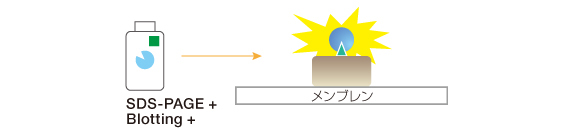
HiBiT付加タンパク質を発現させた細胞を溶解し、SDS-PAGE泳動、メンブレン転写する。メンブレンにLgBiTおよびNanoLuc®基質を含む試薬を添加し、目的タンパク質をバンドとして検出する。
- 試薬を1回添加するだけのシンプルな手順
- 30分以内に実験完了
- 抗体不要・特異的: シグナルはHiBiTとLgBiTの結合でのみ検出
→非特異的バンドは検出されない
-
生細胞の膜たんぱく質、分泌タンパク質の定量 <HiBiT Extracellular Detection>
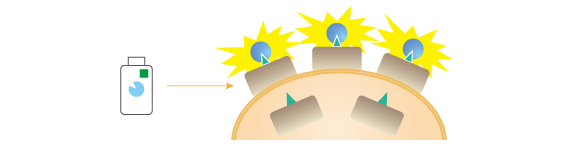
HiBiT を付加したタンパク質として発現した膜タンパク質・分泌タンパク質等を細胞を生かしたまま定量する。
検出試薬に含まれるLgBiT は細胞膜非透過性なので細胞表面のタンパク質または細胞外への分泌タンパク質のみ検出できる。シンプルな添加・検出フォーマットにより、受容体の細胞内移行、受容体のリサイクリング、タンパク質分泌、細胞膜移行をリアルタイムで測定可能。
アプリケーション例- GPCR受容体の生細胞での内在化定量
-
その他
細胞内タンパク質会合による細胞融合、ウイルス感染の検出 <HiBiT Intercellular Detection>
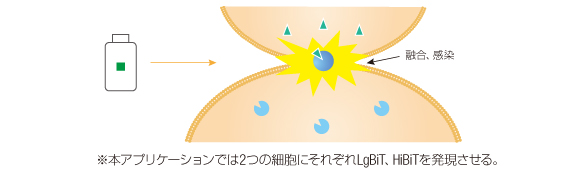
2種類の細胞あるいは細胞とウイルスなどの組み合わせで、それぞれの細胞にLgBiT、HiBiTを発現させ、それらが融合すると自発的に会合し、基質添加により発光定量する。挿入配列の長さに制限のあるウイルスゲノムやCRISPR / Cas9によるゲノム編集に応用することにより、内在性ゲノム、ウイルスゲノム中の遺伝子のマーカーとして利用可能。
アプリケーション例- ウイルス感染あるいは複製 (極小HiBiTがウイルスゲノムへの挿入に好適)
- 細胞融合
- アクセプター細胞へのExosomeデリバリー
- 生細胞での細胞内タンパク質の定量
サイテーション
HiBiT
※ 論文タイトルをクリックするとPubMedに移動します横スクロールできます→
| タイトル | 著者 | ジャーナル名 |
|---|---|---|
Application of a novel HiBiT peptide tag for monitoring ATF4 protein expression in Neuro2a cells. |
Oh-Hashi K, Furuta E, Fujimura K, Hirata Y. | Biochem Biophys Rep. 2017 Aug 15;12:40-45. |
| Sasaki M, Anindita PD, Phongphaew W, Carr M, Kobayashi S, Orba Y, Sawa H. | Virus Res. 2017 Oct 23. | |
CRISPR-Mediated Tagging of Endogenous Proteins with a Luminescent Peptide. |
Schwinn MK, Machleidt T, Zimmerman K, Eggers CT, Dixon AS, Hurst R, Hall MP, Encell LP, Binkowski BF, Wood KV. | ACS Chem Biol. 2017 Sep 21. |
Regions of MRAP2 required for the inhibition of orexin and prokineticin receptor signaling. |
Rouault AAJ, Lee AA, Sebag JA. | Biochim Biophys Acta. 2017 Dec;1864(12):2322-2329. |
Characterization of recombinant Flaviviridae viruses possessing a small reporter-tag |
Tamura T, Fukuhara T, Uchida T, Ono C, Mori H, Sato A, Fauzyah Y, Okamoto T, Kurosu T, Setoh YX, Imamura M, Tautz N, Sakoda Y, Khromykh AA, Chayama K, Matsuura Y. | J Virol. 2017 Nov 1. |
| Eric J Aird, Klaus N Lovendahl, Amber St. Martin, Reuben S Harris, Wendy R Gordon | Communications Biologyvolume 1, Article number: 54 (2018) | |
Quantitative Live-Cell Kinetic Degradation and Mechanistic Profiling of PROTAC Mode of Action |
Riching KM, Mahan S, Corona CR, McDougall M, Vasta JD, Robers MB, Urh M, Daniels DL | ACS Chem Biol. 2018 Sep 21;13(9):2758-2770. doi: 10.1021/acschembio.8b00692 |
| Charles Y. Wanga , Nisha Patela , Wei-Yun Wholeyb , and Suzanne Dawida | PNAS June 19, 2018 115 (25) E5776-E5785 | |
| Deshani C. Ranawakage, Takuya Takada & Yusuke Kamachi | Scientific Reports, 9: 6895 (2019) | |
A Simple and Scalable Strategy for Analysis of Endogenous Protein Dynamics |
Marie K. Schwinn, Leta S. Steffen, Kris Zimmerman, Keith V. Wood & Thomas Machleidt | Scientific Reports 2020, Jun 2;10(1):8953 |
横スクロールできます→