SLAS2024 ポスター情報
プロメガテクノロジー発表ポスター集
DELs to NanoBRET Probes: Enabling Cell-Active Target Engagement
NanoBRET Probeを用いた、生細胞におけるDELスクリーニング評価
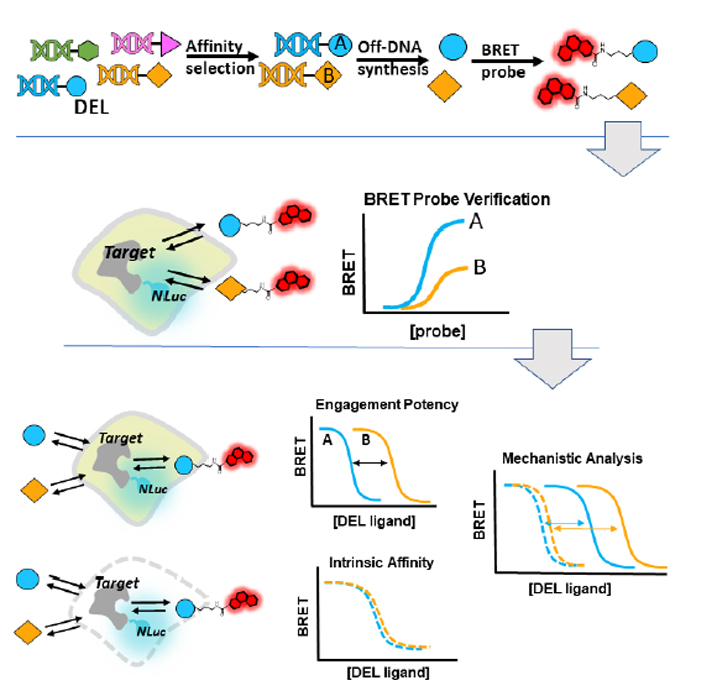
DNA-Encoded Library(DEL)は創薬研究の初期段階において、ヒット化合物を発見するための一般的な手法として用いられています。(DNA-Encoded Library(DEL)についてはこちらをご覧ください)
プロメガではNanoBRET Technologyを応用し、生細胞におけるDELスクリーニング後の評価系を開発しました。DELProbe をNanoBRET Probe に変換することで、生細胞における化合物-標的タンパク質間相互作用の評価が可能となります。
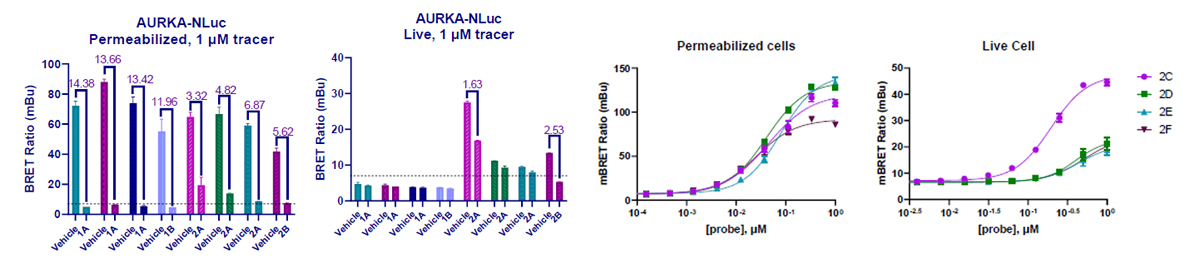
ポスターでは、Aurora kinase Aを対象に膜透過処理・未処理(生細胞)条件下でDELスクリーニングを実施し、化合物と標的タンパク質間の相互作用を評価しています(右図)。
また、BRD4およびIDO1を対象とした評価結果も記載しています。
製品例
- NanoBRET 590-SE (型番:N5000)
- NanoBRET 590-PEG(04)-SE (型番:N5200)
- NanoBRET 590-C4-SE (型番:N5300)
- NanoBRET 590-C3-Azide (型番:N5100)
- Intracellular TE Nano-Glo Substrate/Inhibitor(型番:N2160)
論文事例
DELs enable the development of BRET probes for target engagement studies in cells
Cell Chem Biol. 2023 Jul 5;S2451-9456(23)00197-6
Homogeneous Luminescent Immunoassays for Proliferation and Senescence
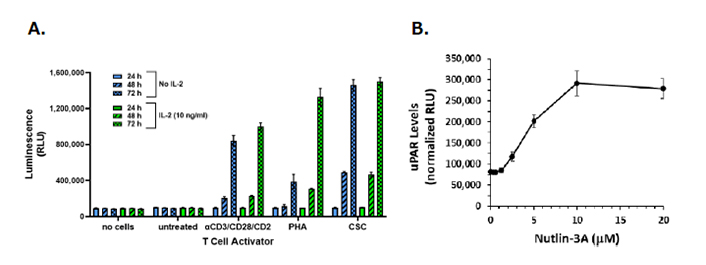
Lumit Immunoassay による増殖因子(Ki-67)および老化マーカー(uPAR)の評価
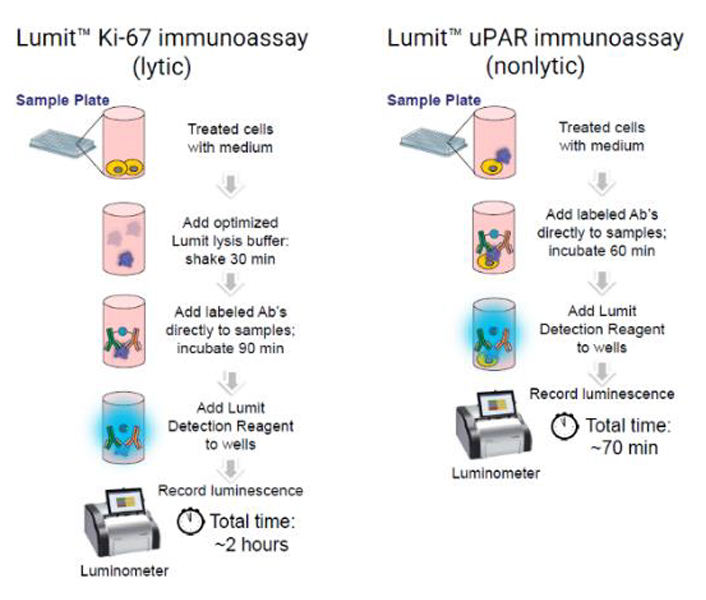
Ki-67は細胞増殖マーカーとして使用される核タンパク質です。細胞周期の全活動期に発現しますが、休止期には存在しません。Ki-67は癌細胞では発現が増加し、老化細胞では発現しないことが知られています。 uPAR (Urokinase plasminogen activator surface receptor) は炎症や腫瘍の進行に関与することが知られ、近年、細胞老化の進行に伴い発現が増加する事が報告されています。Ki-67およびuPARは様々な疾患・病態に関与しており、病態メカニズムや創薬研究における重要な評価因子です。 プロメガではLumit Immunoassay技術を利用し、Ki-67およびuPARを簡単・迅速・高感度に測定可能なアッセイを開発しました。
Lumit Immunoassayは洗浄ステップのないシンプルなプロトコルで、短時間(約2時間)で標的タンパク質を測定できます。また、発光法のためダイナミックレンジが広く、感度良く検出可能です。
本ポスターでは、下記のアプリケーション事例が示されています。
- CD8+T細胞の活性化における細胞増殖・Ki-67発現の評価(下図A) 異なるドナー由来CD8+T細胞でのKi-67発現の比較
- HCT116大腸癌細胞株における増殖阻害剤添加時の Ki-67発現評価
- U937マクロファージ様細胞株におけるPMA添加時の uPAR発現評価
- HCT116大腸癌細胞株における老化誘導に伴うuPAR発現評価 (下図B)
A NanoBRET Target Engagement Assay for querying domain selectivity at full-length Polymerase Theta in live cells
NanoBRETターゲットエンゲージメントアッセイを利用した、生細胞内の全長ポリメラーゼシータにおけるドメイン選択性の解析
NanoBRET™ Target Engagement Assayは、NanoLuc® ルシフェラーゼと蛍光性NanoBRET™トレーサーの近接による、生物発光共鳴エネルギー移動(BRET)を利用するアッセイです。生細胞における標的タンパク質と小分子との結合を、発光測定で検出します(下図)。
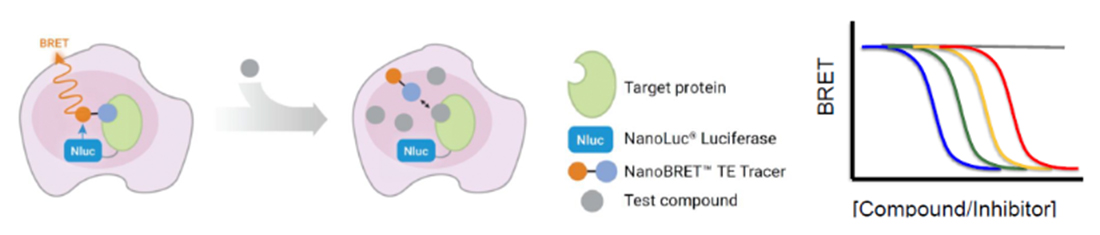
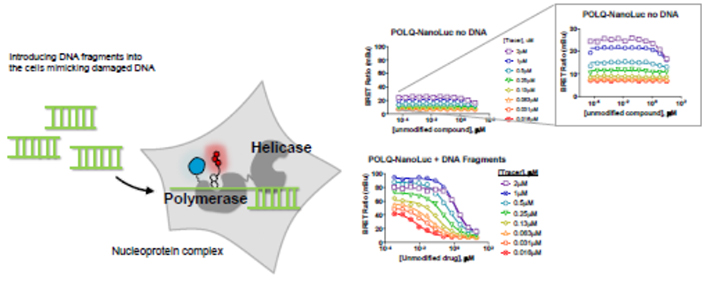
本ポスターでは、ポリメラーゼとヘリカーゼの両ドメインを有するポリメラーゼθをターゲットにした、新たなNanoBRET™ Assayを提示しています。
ポリメラーゼドメインに特異的に結合するプローブとDNA断片を生細胞に導入する手法により、シグナルが増強されました(右図)。
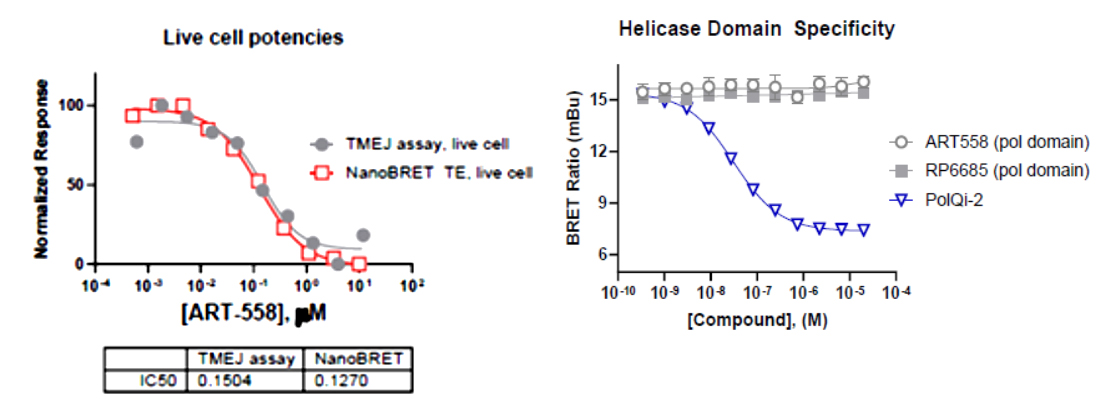
NanoBRETアッセイの結果はTMEJアッセイと相関することが確認されました(下図左)。またヘリカーゼドメインに特異的に結合するプローブも開発されました(下図右)。これらを組み合わせることで、生細胞内のポリメラーゼθの作用メカニズムの詳細な解析が可能となります。
キーワード
組換修復欠損BRCA1/2変異との合成致死
製品例
NanoBRET® 590 Dyes
NanoBRET™ Target Engagement Assayの詳細は、こちらをご確認ください。
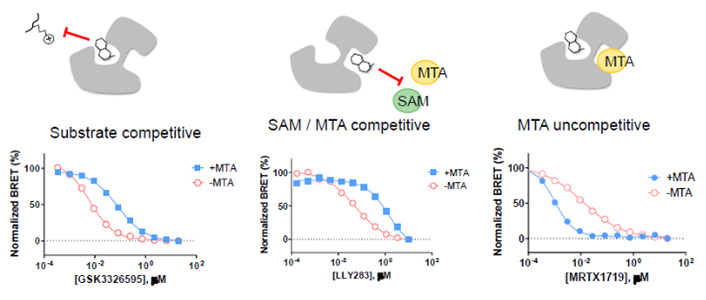
A live cell PRMT5 NanoBRET™ Target Engagement Assay querying competitive and uncompetitive modes of inhibition
NanoBRET™ Target Engagement Assayを用いた生細胞の選択的薬剤結合評価アッセイ
NanoBRET™ Target Engagement Assayでは、標的タンパク質の結合ポケットに対応する細胞膜透過性蛍光プローブを用いることにより、生細胞内にて標的箇所結合薬剤との競合評価を行うことが可能です。
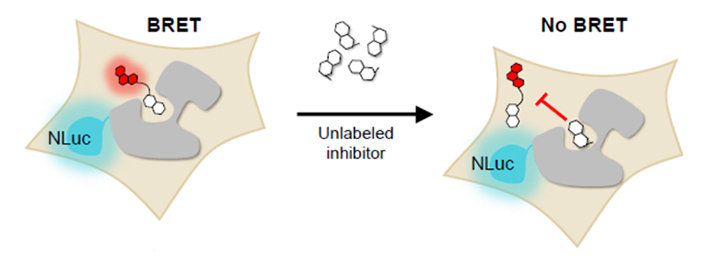
このポスターでは、PRMT5を対象に酵素・補酵素それぞれの活性ポケットへ反応する蛍光プローブを用いて、各薬剤における競合評価を行っています。
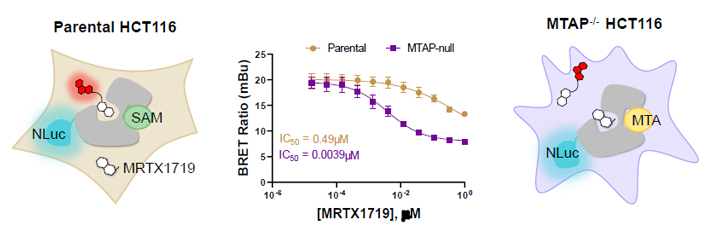
下図は、正常細胞とMTAP(-)細胞にて、補酵素MTA結合のPRMT5特異的に結合する非競合的阻害薬であるMRTX1719を用いて、細胞内PRMT5補酵素結合状態における差を評価しています。
<詳細情報>
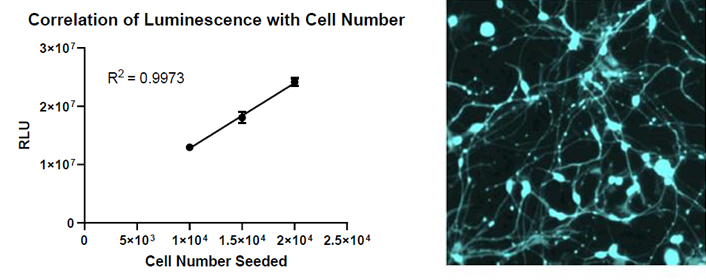
Reporter Systems for Live Imaging of iPSC-Derived Neuronal Cells.
レポーターシステム を用いたiPS細胞由来神経細胞でのLive Imaging
生細胞イメージングを行うには、以下の蛍光色素が必要です。
- 細胞へのダメージを最小限に抑える
- 長時間に渡って蛍光が持続する
- 明るく安定性も高い
本ポスターでは、プロメガが有するHaloTag® 、NanoLuc®の両テクノロジーを駆使し、iPS細胞由来神経細胞におけるレポーターアッセイを紹介しています。
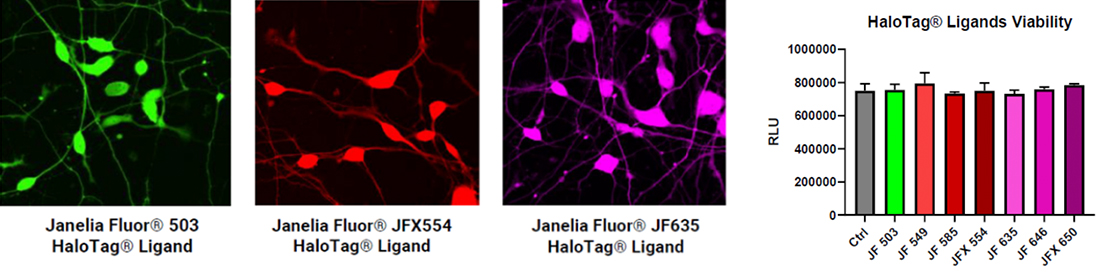
HaloTag®テクノロジーでは、目的・用途に応じてリガンドの選択が可能です。3種の高輝度蛍光リガンドを使用した場合(下図左)、いずれも48時間後の生存性アッセイで細胞毒性がないことを検証済みです(下図右)。
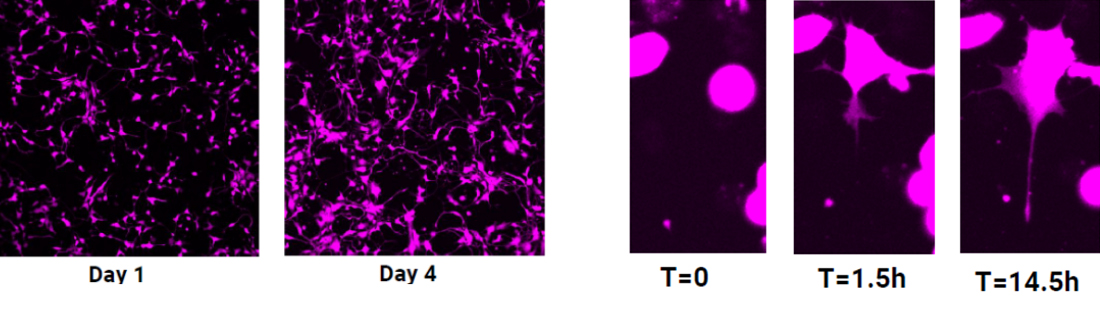
HaloTag® テクノロジーでは、ライブイメージング(下図左)やタイムラプスイメージング(下図右)も可能です。
HaloTag®とNanoLuc® ルシフェラーゼを組み合わせることで、生きた神経細胞での発光による定量・イメージングも可能になりました。
First-in-Class Selectivity System for Quantitative Kinase Inhibitor Profiling in Live Cells Against 192 Kinases
新薬選別のために有用な、生細胞で実施する192キナーゼに対する阻害プロファイル作製
NanoBRET™ TE テクノロジーは、NanoBRET™ を利用した、生細胞で実施可能な阻害剤をはじめとする化合物結合アッセイフォーマットです。結合には細胞内環境の影響があり、in vitroと細胞内で顕著な違いが生じます。このためin vitro反応では細胞内用では有用でない多数の候補化合物もヒットし、細胞内環境でのみ結合の見られるヒットを見過ごすという問題がありました。これらの解決法となるNanoBRET™ TE テクノロジーを用いて、特にシグナル伝達で重要なキナーゼについては数百のアプリケーションが開発し、化合物について「細胞内で」「横断的に」阻害効果を確認することが可能となりました。これは創薬におけるOff-Target 発見や既存薬転用にも有用です。
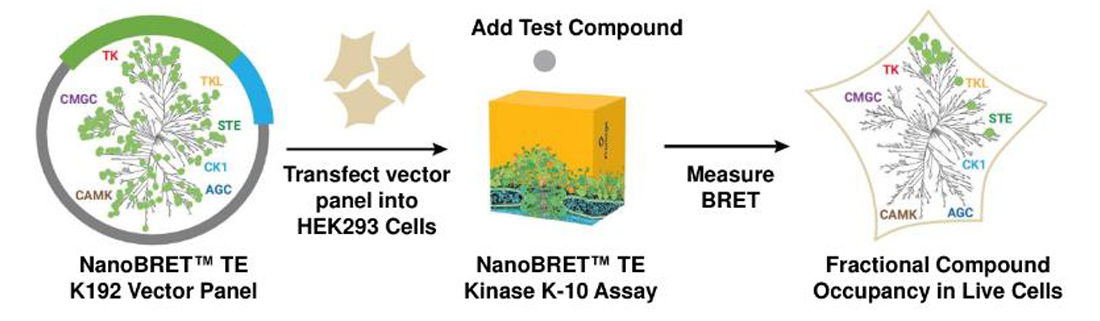
プロメガでは192キナーゼ阻害剤の検証用セットを販売します(上図)。
- アッセイに必要なNanoLucベクターを、便利な96ウェルフォーマットでお届け。
- FuGENE HD トランスフェクションに最適化したProtocol を添付。
- TransfectionNOW HEK293細胞も用いることで、事前・継続培養の手間なく準備1日で開始可能。
- K-10 トレーサー利用した同手順での一括試験が可能。
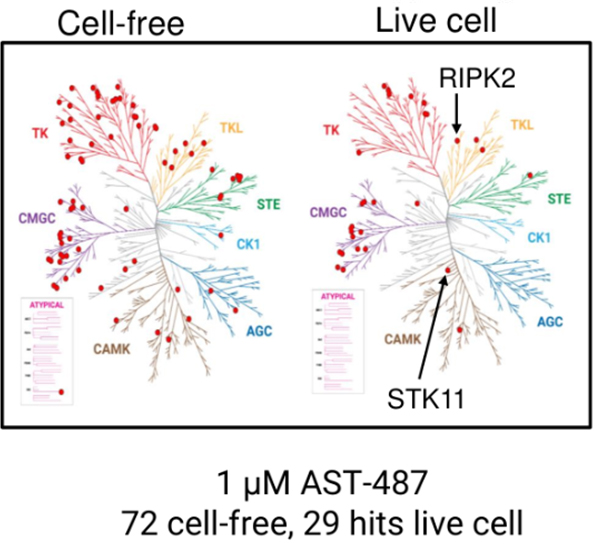
本ポスターでは、細胞内アッセイで実際に細胞内アッセイを細胞外事例と比較し、大きな違いが見られた例を報告しています(右図 樹形図)。
キナーゼ以外にも、プロメガでは各種Target Engagementアプリケーションを開発しております。またTracer 自作に対応する製品もございます。カタログ外アプリケーションも多数ございますので、詳細はお問い合わせください。
製品例
TransfectionNOW™ HEK293 Cell, #NC1001/NC1002
FuGENE® HD, #E2311/E2312 NanoBRET™ TE Intracellular Kinase Assay, K-10, #N2640/N2641/N2840
NanoBRET™ TE Intracellular Kinase Assay, K-10, #N2640/N2641/N2840
Detection and Quantification of Cellular cGAMP using Homogenous HTS Formatted Bioluminescent Assay
HTSフォーマットに対応した、高感度発光アッセイによる細胞内cGAMPの検出と定量化
cyclic GMP-AMP(cGAMP)はサイクリック GMP-AMP 合成酵素 (cGAS) により細胞内で生成されるセカンドメッセンジャーの 1 つです。cGAS が細胞内 DNA を感知すると cGAMP を産生します。cGAMP は細胞内核酸センサー群の 1 つである STING (Stimulator of Interferon Gene) を活性化し、炎症性サイトカインや I 型インターフェロンの産生を強力に誘導し、炎症応答の発生に関与していることが明らかとなっています。
cGAS-cGAMP-STING 経路は、ウイルスなどの外来の微生物に対する防衛策・自然免疫機構として重要視されています。さらに最近では、ウイルス・微生物感染だけでなく、自己免疫疾患やがん、老化、神経変性疾患をはじめとする広範な疾病に関与することが次々と報告されており cGAS-cGAMP-STING 経路は重要な創薬標的となっています。
STING経路を効果的に調節する化合物を探索する開発目標に対して、プロメガでは細胞内および細胞外におけるcGAMP濃度をモニターする発光アッセイ法を開発しました。
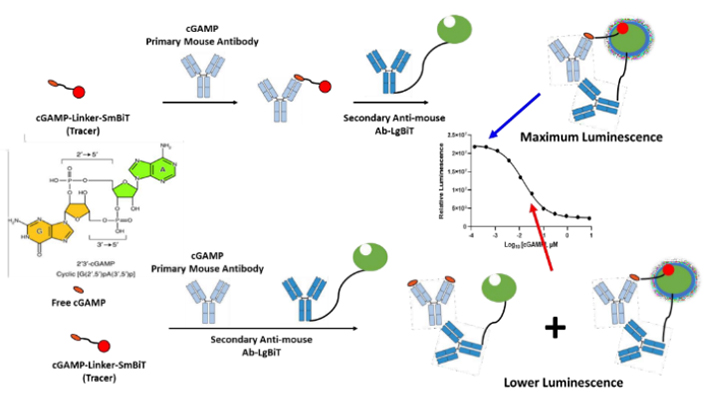
このアッセイ法では、NanoBiTアッセイを応用しています(NanoBiTに関してはこちらをご覧ください)。リンカーを介してcGAMPに結合したSmBiT(cGAMPトレーサー)を、LgBiTに結合した抗cGAMP抗体で認識し、発光基質を添加すると発光シグナルが生じます。この際、反応系の中に遊離cGAMPが存在するとcGAMPトレーサーと競合し、発光シグナルが減少します。(右図)
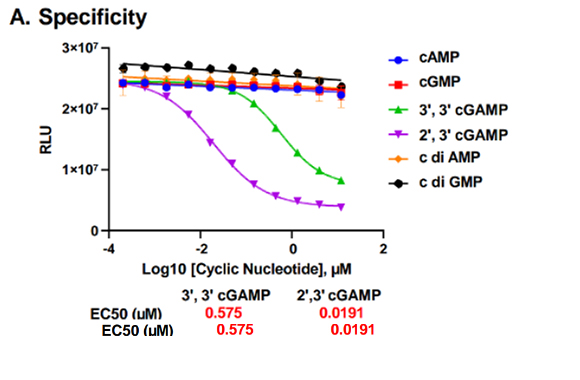
本アッセイのポイント:
- 高感度:10 nM以下のcGAMPを検出可能。 cGAS酵素 10ng以下の反応で生じたcGAMPの検出も可能。
- 細胞内cGMAPの変動評価も可能: dsDNA刺激を用いたcGASの活性化と、cGASレポーターアッセイの活性化で相関を確認。
- 迅速性:1時間未満で測定完了。ホモジニアスアッセイで、HTSにも十分対応可能
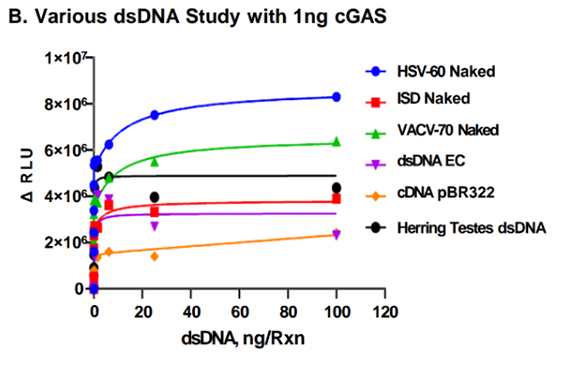
本アッセイはSTING経路のモジュレーターの研究や新規治療薬の探索に有用です。
先行販売品としてご提供が可能です。詳細はプロメガ学術課までお問合せ下さい。
Evaluation of PROTAC- and Molecular Glue- Mediated Degradation of Neosubstrates using CRISPR HiBiT-tagged Cell Lines in a High-Throughput Screening Workflow
HiBiTノックイン細胞株を用いたPROTACおよびMolecular Glue(MG)によるネオ基質分解評価
標的タンパク質分解(TPD)創薬として注目されているPROTACやMolecular Glue(MG)では、標的外の分解を最小限にしながら標的特異性を維持することが求められています。
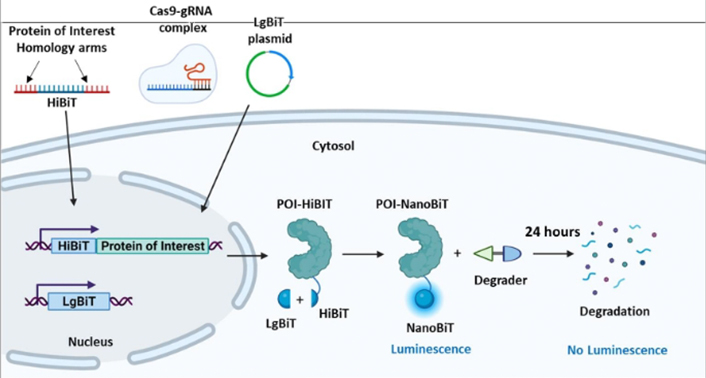
本ポスターでは、代表的なオフターゲットネオ基質6種(CK1α、GSPT1、IKZF1、IKZF2、IKZF3、SALL4)のHiBiT KI細胞株を使用し、市販12種類のPROTACとMGを評価しています。
また、各PROTAC、MGの標的分子との比較から、On-target/Off-target効果のポテンシー比較にも使用できます。これら評価は自動化のシステム構築により、ハイスループットでの評価も可能としています。
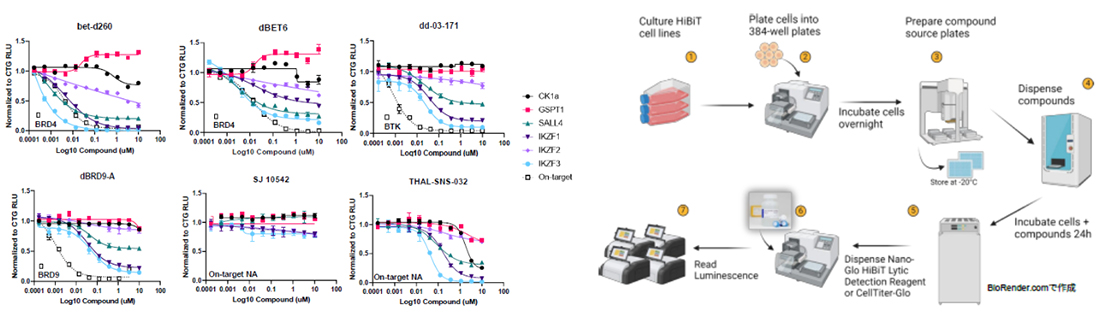
図. 6種のPROTACによるOn-target効果と6種ネオ基質Off-target効果のポテンシー比較
ネオ基質6種のパネル細胞を用いた解析サービス、ならびに各細胞株の個別販売を行っております。詳細ご希望の際はプロメガ学術課へお問い合わせください。