E. coli S30 Extract System
- E. coli S30 Extract Systemを用いた発現系では、鋳型DNAをどのように設計すればよいですか?
-
大腸菌で発現させるためのプロモーターとその下流に適切なRibosomal Binding Site (RBS)を含むように設計してください。大腸菌でよく発現する共通のプロモーターとしては、lacUV5、tac、λPL、lac、trpなどがあります。ベクターを用いて目的タンパクの遺伝子をクローニングする場合は、ベクター配列にある大腸菌発現プロモーターを確認してください。
表2 大腸菌発現プロモーターのコンセンサス配列
プロモーター -35 region -10 region コンセンサス配列 (1) TTGACA TATAAT lacUV5 (2) TTTACA TACAATG tac (2) TTTACA TTTAATG λPL(2) TTGACA ATAAT lac (2) TTTACA TATGTT trp (2) TTGACA TTAACTA 開始コドンから適切な間隔をあけて位置しているRBSは、E. coliの発現系で翻訳を開始させるために必要です。例えば、pF1A(K) T7 Flexi® Vector(C8441、C8451)のRBSは、Shine-Dalgarno(SD)配列の「AGGAG」(3)を用いています。
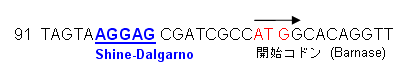
E. coli T7 S30 System for Circular DNAを使用し、T7 promoterの配列を介した転写反応を実施する場合は、例として、以下のとおりに鋳型DNAの配列設計してください。
<参考>
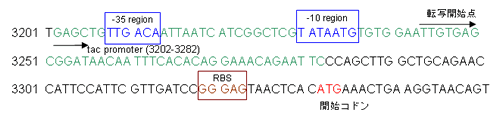
大腸菌発現ベクターとして販売されている、PinPointTM Xa-1 Vector(V2031)のtacプロモーター領域から開始コドンの間の配列を示します。
参考文献
(1)Nucleic Acids Res., 17, 2933?2945, 1989
(2)JBC., 260, 3539-3541,1985
(3) Nature 254, 34, 1975
お電話でのお問い合わせ
製品詳細・学術的なお問い合わせ専用ダイヤル(受付時間:平日9:00~17:00)
TEL 03-3669-7980
